Lecture:Phân loại học phân tử/Nhóm 3/Bài 3
Tuần 3[sửa]
- 16S.Tri.fasta: Cut off at 1800 score, 2 sequences were discarded (HM921188.1, HM921189.1).
- 16S.nonTri.fasta: Cut off at 1800 score, 11 sequences were discarded (AF113639.1, EF016446.1, HM040948.1, GU213804.1, GU213806.1, GU213811.1, GU213824.1, GU213825.1, HM921173.1, HM921185.1, HM921191.1)
- 16S.all.fasta: combined off the two files, using cat. >> ERROR: sequence with the same names!
Retrive for all sequences:
Received lines: 560
Rejected lines: 0
Removed duplicates: 185
Passed to Entrez: 375
Retrieve records for 375 UID(s)
Đã thực hiện download lại với allsequences (16S.Tri và 16S.nonTri (từ Thọ) kết hợp, loại bỏ các số hiệu lặp): Kết quả giữ lại 362 sequences trong 375 sequences khi vẫn giữ cut off tại 1800.
CLUSTALW2 for Tri [1] (FINAL)
CLUSTALW2 for all [2]
Kết quả chạy rất nhanh (đã xuất hiện) với cả hai file (trên đó có hỗ trợ đồ họa). Toàn bộ qui trình và file tạm thời được lưu tại đây [3]
Theo gợi ý của anh Hiếu, score hạ xuống 1600 cho all sequences, loại 8 trình tự [4]. Cắt sâu vào 20 nu, loại bỏ thêm 8 trình tự có một trong hai đầu hụt (tổng cộng loại 16 trình tự.)
Trình tự GU213823.1 thể hiện rất đặc biệt: nhiều point mutations hơn các trình tự khác khi quan sát bằng mắt (loại bỏ trình tự này -> tổng cộng loại 17 trình tự.)
Kết quả của all sau khi loại bỏ các trình tự quá sai (17 trình tự) + cắt bỏ 20 nu đầu tiên [5] (FINAL)
Tóm tắt quá trình[sửa]
1. Download và chuẩn bị dữ liệu: Download 16S accession number search từ ncbi cho Tri (34 trình tự) (file 1). Download 100 accession number của trình tự hight score từ blast của ncbi (file 2). Lấy accession number của 16S - not Tri từ file sequences của Thọ (file 3) (326 sequences). Kết hợp file 1 và 2 được file 16S.Tri.txt. Kết hợp file 1, file 2 và file 3 được 16S.all.txt. Dựa trên 2 file đó download các sequence 16S.Tri.fasta và 16S.all.fasta. (Tuy đơn giản nhưng khá dễ nhầm lần lung tung.)
2. Edit tên trình tự bằng script, tạm thời chỉ edit phần đầu (cho nhanh.)
3. Cắt trình tự bằng pairwise algiment với một trình tự tiêu bản. cho ra hai file có đuôi .cut. Quá trình cut thực hiện đối với cả trình tự để xuôi và reverse complement, lấy sắp xếp score cao hơn. Các trình tự reverse hoặc không reverse được report ra màn hình. Kết quả score được vẽ lên biểu đồ, căn cứ vào đó đặt threshold cho score để cắt loại trình tự có score thấp một cách đáng nghi ngờ (report ra screen.) Tổng số trình tự cắt report ra screen. (Thời gian rất thuận lợi: khoảng 3 phút cho 300 trình tự.)
Chi tiết hơn về script: File đầu vào gồm hai file: file tiêu bản (pattern) và toàn bộ các sequence (chứa cả tiêu bản mẫu). R sẽ lấy tiêu bản so sánh với tất cả các sequence, và reverse của các sequence đó, chọn lấy đoạn có score cao nhất. Tiếp đó dựa theo threshold đưa vào (qua quan sát đồ thị) sẽ cắt các trình tự đáng nghi loại bỏ. Tất cả các thao tác cắt và reverse report re screen. File ra xuất ra trong câu lệnh cuối cùng là file đã cắt, reverse sequence, delete extrems.
4. Cho trình tự đã edit lên server, run server. File Tri có thể giữ lại không cần làm gì thêm. File All chưa tốt: tiếp tục edit và chạy lại file all (cắt 20 nu đầu, loại bỏ tất cả các trình tự hụt đầu và hụt đuôi) (có hỗ trợ công cụ cắt trên server). File sau khi đã cắt chạy tương đối nhanh (khoảng 10 phút.)
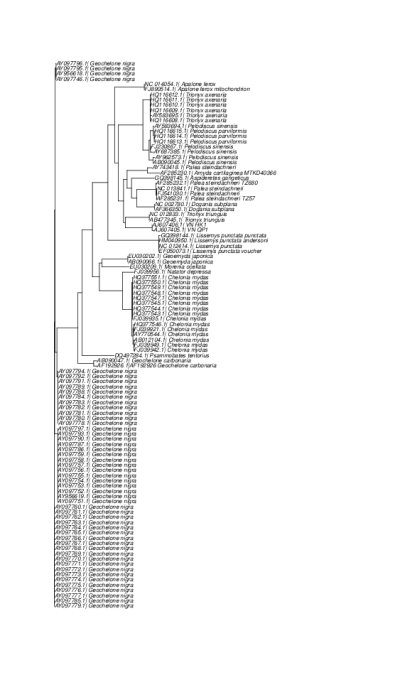
Kết quả clustalw sau khi cắt file + tree + bootstrap [6].
5. Tiếp theo: annotation.
Cây của Tri và all + annotation [7] (Do annotation dày đặc nên khi xuất ra hình nói chung rất khó nhìn với file all.)
Cây của nhóm Tri + annotation