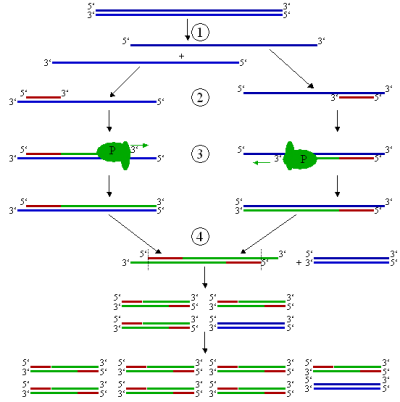
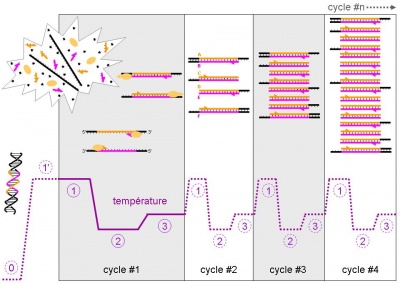
Sách:Cẩm nang SHPT/Chương 18. Kỹ thuật PCR căn bản
PCR là một kỹ thuật phổ biến trong sinh học phân tử nhằm nhân bản (tạo ra nhiều bản sao) một đoạn DNA trong ống nghiệm mô phỏng bộ máy sinh tổng hợp DNA của tế bào sống. Kỹ thuật này được sử dụng rộng rãi trong các nghiên cứu sinh học và y học phục vụ nhiều mục đích khác nhau, như phát hiện các bệnh di truyền, nhận dạng vân tay DNA, chuẩn đoán những bệnh nhiễm trùng, tách dòng gene, và xác định huyết thống.
Các lưu ý
Thủ thuật thiết kế primer
Thành phần phản ứng
Enzyme
- Các đặc tính enzyme DNA polymerase và định nghĩa đơn vị (unit) của mỗi loại (hãng) sản xuất là khác nhau. Cần kiểm tra thông tin trên các tờ hướng dẫn đi kèm hóa chất.
- Lượng enzyme cần thiết là phụ thuộc vào lượng DNA template và kích thước phản ứng PCR. Tuy nhiên, thành phần này chỉ nên hiệu chỉnh khi 1) sản phẩm PCR lớn hơn 5kb; 2) có khả năng hoạt tính của enzyme bị giảm sút do thiếu sót ở khâu bảo quản; 3) đã tiến hành hiệu chỉnh bằng nhiều cách nhưng sản phẩm PCR vẫn thấp hoặc không có gì.
- Không nên tăng enzyme quá 2U (5µl) trên 50 µl tổng thể tích phản ứng vì các chất trong dung dịch bảo quản enzyme có thể gây ức chế phản ứng
- Việc thay đổi loại DNA polymerase có thể làm thay đổi hiệu suất của phản ứng PCR.
- Khi tính toán chiến lược cloning bằng hệ thống TA vector, cần lưu ý DNA polymerase có đặc tính tạo đầu treo A ở đầu 3' hay không. Nếu không thì cần phản ứng bổ sung sử dụng Klenow và ATP.
- Tính chính xác của enzyme DNA polymerase phụ thuộc vào hoạt tính 3´→5´ exonuclease. Tùy thuộc mục đích thí nghiệm mà cần lựa chọn loại DNA polymerase có hay không có hoạt tính này.
Buffers
- Hiệu suất, tính chính xác của enzyme DNA polymerase phụ thuộc nhiều vào hệ buffer sử dụng. Thông thường nên sử dụng đúng loại buffer tương ứng với enzyme do nhà sản xuất khuyến cáo.
- Một số buffer chứa chất hoạt động bề mặt có thể ảnh hưởng đến các phản ứng microarray hoặc DHPLC.
- Nhiều loại buffer đã có sẵn một lượng nhất định ion Mg2+ nên cần lưu ý khi tối ưu hóa nồng độ Mg2+
Nồng độ Mg2+ và dNTP
- Việc tăng nồng độ ion Mg2+ có thể làm xuất hiện thêm các sản phẩm không đặc hiệu. Tuy nhiên nếu thiếu ion Mg2+ thì dẫn đến lượng sản phẩm PCR thấp.
- Nồng độ ion Mg2+ tối ưu phụ thuộc vào loại DNA polymerase, loại DNA template và thành phần buffer (một số buffer thương mại đã có sẵn 1 lượng nhất định ion Mg2+).
- Dò tìm nồng độ Mg2+ tối ưu thông thường bằng gradien nồng độ từ 0.5 đến 2 mM với các khoảng cách mỗi bước là 0.2 mM.
- dNTPs là loại hóa chất kém bền cần lưu ý khi sử dụng và bảo quản. Nên chia nhỏ lượng dNTPs để giảm số lần làm tan, sử dụng lại.
- Lượng dNTP tối ưu là 200 µM of mỗi loại dNTP. Việc tăng dNTP không làm tăng đáng kể lượng sản phẩm PCR so với việc hiệu chỉnh các yếu tố khác.
Template và Primers
- Nồng độ tối ưu DNA template là từ 1) đối với các mẫu DNA tương đối đồng nhất (plasmid, lambda hoặc BAC DNA): 1 pg - 10 ng / 50 µl tổng thể tích phản ứng; 2) đối với mẫu DNA genome thì từ 20 - 500 ng/50 µl tổng thể tích.
- Nồng độ primer thường tối ưu ở 10 mM mỗi loại.
Chất khác
- Đối với các DNA template lớn, giàu GC thì có thể sử dụng các chất biến tính như DMSO, formamide, glycerol, and betaine trong thành phần PCR. Tuy nhiên, phải kiểm tra độ tương thích đối với enzyme DNA polymerase.
- Tăng lượng DMSO trong PCR sẽ cần giảm nhiệt bắt mồi xuống.
- Một số buffer đã có chứa sẵn loading buffer để có thể điện di ngay sau PCR. Tuy nhiên, độ nhạy của PCR với những buffer này không cao và sản phẩm sau đó ko dùng trực tiếp cho đọc trình tự mà cần phải tinh sạch qua cột.
- Đối với các máy luân nhiệt (thermocycler) không có gia nhiệt trên nắp thì nên bổ xung mineral oil để tránh bay hơi mẫu.
Chu trình nhiệt
Biến tính
- Việc tăng nhiệt độ hoặc kéo dài thời gian biến tính có thể làm tăng tính đặc hiệu của PCR nhưng làm giảm tuổi thọ của enzyme DNA polymerase.
- Đối với những enzyme DNA polymerase bền nhiệt thường nên sử dụng nhiệt độ biến tính 95°C-98°C. Bao gồm bước biến tính dài lúc khởi đầu và một bước biến tính ngắn trước từng chu kỳ nhiệt.
- Đối với các genome lớn như thực vật thường sử dụng bước biến tính dài đến 5 min. Để làm giảm ảnh hướng đối với enzyme thì có thể bổ sung enzyme DNA polymerase sau khi biến tính ban đầu.
- Khi đã tối ưu PCR thì cần giảm tối thiểu thời gian biến tính mỗi chu kỳ để tăng lượng sản phẩm. Thông thường từ 10s đến 30s.
- Tốc độ gia nhiệt và chế độ điều khiển nhiệt độ ở mỗi hệ thống luân nhiệt (PCR machine, thermocylcer) là khác nhau phụ thuộc hãng sản xuất. Một số hệ thống cho phép thay đổi thông số này khi tối ưu PCR.
Bắt mồi
- Đối với primer dài hơn 22 nucleotide, nhiệt độ bắt mồi thường hoạt động ở Tm thấp nhất + 3°C.
- Đối với primer nhỏ hơn 22 nucleotide, nhiệt độ bắt mồi thường thấp hơn Tm thấp nhất, có thể dùng grandient nhiệt độ (touchdown PCR) để tìm nhiệt độ bắt mồi tối ưu (khoảng cách nhiệt ở mỗi chu kỳ thường là 0.5°C).
- Đối với các cặp Tm cao hoặc tự bắt cặp, có loop thì có thể khắc phục bằng PCR 2 bước (95°C và 68°C).
Kéo dài
- Nhiệt độ kéo dài thường tiến hành ở 72°C tuy nhiên, hoạt tính 5'-3' DNA polymerase vẫn xảy ra ở nhiệt độ 68°C
- Thời gian kéo dài phụ thuộc hoạt độ enzyme DNA polymerase, chiều dài sản phẩm và loại template. 1) Đối với DNA đồng nhất (plasmid, lambda hoặc BAC DNA) thì tốc độ có thể đạt tới 15s/kb; 2) đối với DNA genome lớn, phức tạp thì tốc độ thường 30s-1min/kb.
Động học của phản ứng
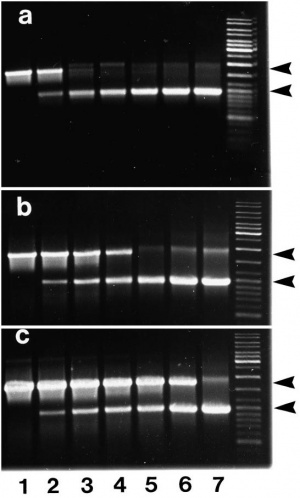
Lượng sản phẩm PCR được tổng hợp nên sau mỗi chu kỳ thường theo 1 dạng đường chuẩn gồm 3 giai đoạn: 1) tăng trưởng mũ, 2) tăng tuyến tính, 3) bão hòa (lượng sản phẩm tăng không đáng kể hoặc thậm chí giảm). Theo động học của 1 phản ứng enzyme thông thường, nhiều nguyên nhân được giả định như 1) sử dụng hết cơ chất (dNTPs hoặc primers); 2) giới hạn nồng độ enzyme hoặc enzyme dần bị bất hoạt do nhiệt; 3) việc tăng dần nồng độ pyrophosphate gây ức chế hoạt động enzyme; 4) hiện tượng tự bắt cặp của các sản phẩm PCR đặc hiệu khi nồng độ sản phẩm cao hơn 10-8M; 5) hiệu quả bước nhiệt biến tính bị giảm dần sau mỗi chu kỳ và 6) hoạt tính 5-3 exonuclease của Taq DNA polymerase phân hủy một phần sản phẩm PCR. Tuy nhiên, thí nghiệm của Peter Kainz (BBA, 2000) chỉ ra nồng độ DNA nói chung (gồm cả sản phẩm PCR và DNA lẫn tạp) và lượng DNA polymearase tự do có mặt là yếu tố chính quyết định điểm bão hòa sản phẩm trong PCR.
Khi Kainz tăng nồng độ DNA đầu vào (nhưng giữ nguyên số DNA khuôn thực) bằng cách bổ sung 1 lượng khác nhau (từ 200ng - 2ug) của một đoạn DNA lẫn tạp (kích thước nhỏ hơn kích thước sản phẩm PCR), PCR bị ức chế bởi DNA ngoại lai và có sự phu thuộc rõ ràng vào nồng độ của DNA ban đầu. Bằng cách bổ sung thêm lượng DNA polymearase có mặt trong phản ứng có thể giảm thiểu sự ức chế này (xem hình 1). Điều đó cho thấy hoạt động của DNA polymearase phụ thuộc chặt chẽ vào nồng độ DNA có mặt trong ống thí nghiệm (bất kể là sản phẩm đặc hiệu hay DNA lẫn tạp). Điều này được tái khẳng định khi thử nghiệm khả năng kéo dài của DNA polymerase (hairpin extension assay) ở PCR sau một số lượng chu kỳ nhất định. Ở thí nghiệm của Kainz, hoạt tính DNA polymerase bị ức chế 1 phần sau 20 đến 25 chu kỳ và gần như toàn bộ sau 30 - 40 chu kỳ. Nói cách khác, hiện tượng bão hòa sản phẩm PCR là do thiếu lượng DNA polymerase tự do cần thiết cho mọi chặc sao chép.
Vai trò của các thành phần khác trong PCR được xác định bằng thí nghiệm PCR 2 cấp. Một PCR với 25 chu kỳ đầu tiên trong đó thiếu đi một số thành phần chính 1) chỉ có dNTP và DNA polymerase; 2) đủ các thành phần; 3) dNTP, DNA polymerase và primer (thiếu DNA khuôn). Sau đó các ống PCR này được bổ sung thành phần còn thiếu 1) primer và DNA khuôn; 3) DNA khuôn rồi chạy tiếp 25 chu kỳ nữa. Trong cả 3 trường hợp thì lượng PCR tạo ra sau cùng đều có thể phát hiện được (xem hình 2). Nhưng lượng sản phẩm thấp nhất (ít hơn 1/10 sản phẩm của các lô khác) là của lô số 3 khi hoạt động DNA polymerase bị ức chế bởi do sản phẩm không đặc hiệu (primer artefacts) được tạo ra từ 25 chu kỳ đầu. Tác dụng tiêu cực này có thể khắc phục khi tăng lượng DNA polymerase đầu vào. Ngoài ra, sản phẩm tạo ra sau 50 chu kỳ (lô số 2) ít hơn đáng kể so với sản phẩm tạo ra từ 25 chu kỳ sau (lô số 1) dù DNA polymerase và dNTP chịu nhiệt của cả 50 chu kỳ.
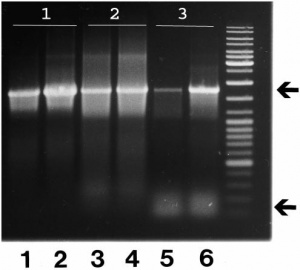
Với những tính toán của mình trong nghiên cứu đó, Kainz cho rằng khi số phần tử DNA tổng số vượt gấp 30 lần số phần tử DNA polymerase là lúc xảy ra sự bão hòa sản phẩm PCR. Và rằng việc tăng lượng DNA polymerase (hơn 1U / 50ul phản ứng) không được khuyên dùng vì sẽ tạo ra lượng lớn sản phẩm không đặc hiệu khác nhau.
Giải pháp tối ưu hóa
Không có sản phẩm hoặc lượng sản phẩm quá thấp
- Lặp lại thí nghiệm đề phòng lỗi thao tác.
- Sử dụng ống dNTP mới, có chất lượng đảm bảo.
- Tăng lượng DNA template, điện di kiểm tra lại nồng độ DNA template.
- Tinh sạch DNA template bằng chiết phenol, tủa cồn hoặc kit tinh sạch hoặc thôi gel. Nếu có thể cần tiến hành tách chiết mới DNA template bằng các phương pháp tách chiết khác nhau.
- Tăng thời gian kéo dài đối với các sản phẩm PCR trên 0,5 kb.
- Tăng số chu kỳ PCR lên 35, 40.
- Hạ thấp nhiệt độ bắt mồi xuống 1 đến 2°C. Hoặc sử dụng touchdown PCR.
- Dùng PCR đánh bắt với nhiệt độ bắt mồi khá thấp (~45°C) trong 10 chu kỳ đầu và nâng cao nhiệt độ đến Tm thấp nhất ở 20 chu kỳ tiếp theo.
- Tăng nhiệt độ biến tính (tối đa 98°C) và điều chỉnh thời gian biến tính DNA đối với các mẫu DNA của genome lớn hoặc khi khuyếch đại các đoạn lặp SSR.
- Kiểm tra các điều kiện của primer, thiết kế cặp primer mới để chạy nestPCR.
- Tăng nồng độ enzyme DNA polymerase
- Thay đổi loại enzyme DNA polymerase và hệ buffer
Vệt smear kích thước lớn hơn sản phẩm mong muốn
- Giảm nồng độ enzyme DNA polymerase
- Rút ngắn thời gian kéo dài
- Giảm số chu kỳ nhiệt PCR xuống tối đa 25.
- Tăng nhiệt độ bắt mồi lên 2°C hoặc thử với PCR 2 giai đoạn hoặc nestPCR
- Thay đổi nhiệt độ biến tính.
- Thử chạy gradien nồng độ Mg2+
- Hạ thấp nồng độ primer.
- Đối với trường hợp vệt smear thấp hơn băng mong muốn thường được tối ưu bằng cách tăng thời gian kéo dài cuối cùng ở 72°C từ 8 đến 10 min.
Băng sản phẩm phụ rõ nét với kích thước thấp hơn sản phẩm mong muốn
- Tăng nhiệt độ bắt mồi 2°C và/hoặc tăng thời gian bắt mồi
- Tăng thời gian kéo dài thêm 30s.
- Giảm nồng độ DNA polymerase.
- Tối ưu hóa nồng độ Mg2+.
- Tinh sạch DNA template bằng thôi gel. Hoặc thủy phân DNA bằng một số RE trước khi dùng làm template (hiệu quả khi băng sản phẩm phụ kích thước khá lớn >= 1kb).
- Giảm nồng độ primer.
- Thiết kế cặp primer mới.
Liên kết hữu ích
- PCR application manual của Roche
- PCR troubleshooting từ Med. Yale Uni.
- PCR Protocols trên Protocol-online
- DNA and General PCR Methods trên Biotechniques
- The PCR plateau phase – towards an understanding of its limitations, Peter Kainz (2000) Biochimica et Biophysica Acta (BBA) - Gene Structure and Expression 1494 (1-2), 23-27
- Bảng tính giá trị Tm của oligo primer trên Applied Biosystems
Bài cùng thể loại
- Làm sạch nhanh sản phẩm PCR để giải trình tự
- Protocol tách protein từ inclusion bodies trong điều kiện biến tính
- Lựa chọn hệ biểu hiện để sản xuất protein ở vi khuẩn
Liên kết đến đây
- Cẩm nang cho nhà sinh học phân tử
- Quy trình Công nghệ
- Bản mẫu:Quy trình mới
- Lựa chọn hệ biểu hiện để sản xuất protein ở vi khuẩn
- Sinh học phân tử
- Thành viên:Cao Xuân Hiếu/Note: Protocol tách protein từ inclusion bodies trong điều kiện biến tính
- Thành viên:Cao Xuân Hiếu/Note: Làm sạch nhanh sản phẩm PCR để giải trình tự