Tạo hình cấu trúc protein
Mục lục
Mô hình nhiệt động học[sửa]
The following points are made by Jeffery W. Kelly (Nature 2006 442:255):
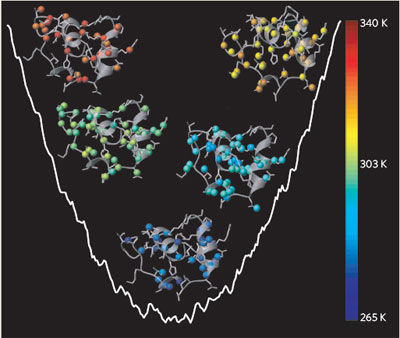
1) Vì rằng cấu trúc không gian 3 chiều là yếu tố quyết định chức năng của mỗi protein và đòi hỏi quá trình tạo hình chính xác. Protein sử dụng một mạng lưới các tương tác hóa học yếu, không đồng hóa trị để đạt đến trạng thái cấu hình hoàn hảo [1]. Trước đây, những nhà khoa học cho rằng quá trình tạo hình protein là một quá trình tương tác hoàn hảo - protein chỉ tồn tại ở 2 trạng thái là chưa tạo hình và tạo hình hoàn hảo. Mô hình "tất cả / không" này thuận tiện cho việc chuyển đổi các dữ liệu quang học thành nhiệt động học chỉ bằng cách đơn giản là ước tính tỉ lệ phân bố của phân tử cuộn xoắn và không cuộn xoắn. Tuy nhiên mô hình này liệu có đúng trong mọi trường hợp? Muñoz et al [2] đã sử dụng phương pháp cộng hưởng từ hạt nhân (NMR) để theo dõi quá trình biến đổi của một protein với các vòng xoắn alpha có tên là BBL.
2) BBL đã tạo hình theo một mô hình "thung lũng năng lượng" (downhill) - quá trình này cho rằng tồn tại các hàng rào năng lượng cực thấp giữa các trạng thái cuộn xoắn và không cuộn xoắn [3],[4],[5]. Khi một protein đang tạo hình lại bắt gặp những điều kiện không thuận lợi như stress nhiệt thì sẽ làm chúng tháo cuộn xoắn dần dần [6]. Những phát hiện khác [7] cũng ủng hộ giả thuyết này, và cho rằng quá trình tạo hình protein không nhất thiết phải tuân thủ mô hình "tất cả / không" bởi vì việc tháo cuộn xoắn được cho là một quá trình ngược chiều với tạo hình, và nghiên cứu nó sẽ tăng hiểu biết về quá trình tạo hình protein.
3) Mỗi nguyên tử hydro trong protein đều nằm trong một môi trường điện tích đặc trưng được tạo thành bởi các nhóm chức xung quanh. Do đó, mỗi nguyên tử này đều có một tín hiệu riêng trên phổ NMR, và tần số của tín hiệu này thay đổi khi protein này trở nên tháo cuộn xoắn làm môi trường xung quanh nguyên tử thay đổi. Khi môi trường được gia nhiệt để kích thích quá trình tháo cuộn xoắn protein, BBL có thể đạt đến những cấu hình không gian khác nhau và chuyển đổi khá nhanh, do đó tần số NMR của mỗi nguyên tử hydro sẽ là vật chỉ thị cho mức độ tháo cuộn xoắn cục bộ.
4) Muñoz et al [8] đã ghi lại những thay đổi về tần số NMR của 158 nguyên tử hydro của BBL theo sự biến thiên của nhiệt độ để tạo nên 158 đường đồ thị nguyên tử được gọi là đường cong tháo cuộn xoắn. Những đường cong này này là không thể biến thiên 2 chiều. Mà rằng, nhiệt độ trung tâm của những đường biến thiên nhiệt là vào khoảng 60 K. Điều này chứng tỏ, tại bất kỳ nhiệt độ nào, tồn tại một vài nguyên tử hydro ở điều kiện tạo hình hoàn chỉnh và một số còn lại thì ở môi trường chưa hoàn chỉnh. Thực tế này đã bác bỏ giả thuyết rằng BBL chỉ tồn tại 2 trạng thái duy nhất là tạo hình hoàn chỉnh và trạng thái nguyên sơ [9].
Chức năng của các phân tử chaperone[sửa]
R. John Ellis (Nature 2006 442:360)[sửa]
1)
Protein
là
những
phân
tử
thực
thi
các
chức
năng
của
sự
sống,
và
để
tạo
ra
những
protein
với
đầy
đủ
chức
năng,
tế
bào
phải
thực
hiện
những
quá
trình
cực
kỳ
nghiêm
ngặt.
Nhờ
hoạt
động
của
những
siêu
cấu
trúc
gọi
là
ribosome,
những
amino
acid
được
nối
lại
thành
các
chuỗi
polypeptide
không
phân
nhánh.
Mỗi
chuỗi
polypeptide
này
sau
đó
được
cuộn
chặt
để
thành
một
hình
dạng
với
bề
mặt
đặc
trưng
và
cần
thiết
của
mỗi
phân
tử
protein.
Thông
tin
quyết
định
việc
tạo
hình
chính
xác
từng
vị
trí
là
nằm
ở
trình
tự
của
chuỗi
amino
acids,
trình
tự
này
đã
được
dịch
mã
tương
ứng
từ
thông
tin
di
truyền
trên
mRNA
do
đã
được
sao
chép
từ
gene
tương
ứng.
Tuy
nhiên,
để
có
thể
tổng
hợp
một
lượng
protein
đủ
nhanh,
mỗi
phân
tử
mRNA
thường
được
dịch
mã
cùng
một
lúc
bởi
nhiều
ribosome.
Điều
này
dễ
dàng
làm
cho
các
chuỗi
polypeptide
mới
được
cuộn
xoắn
từng
phần
bám
chặt
lẫn
nhau
vì
chúng
nằm
quá
gần
và
có
cấu
trúc
giống
hệt
nhau.
Đống
protein
hổ
lốn
này
không
thể
tiếp
tục
cuốn
xoắn
bình
thường
và
cũng
không
thể
thực
hiện
các
chức
năng
sinh
học
được
nữa.
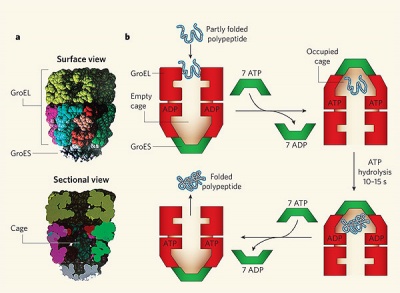
2) Vấn đề cực kỳ phổ biến ở mọi loài sinh vật này đã được giải quyết bởi một nhóm protein có tên là chaperone mà hiện giờ người ta đã phát hiện hơn 50 họ protein khác nhau trong nhóm này [10]. Một họ protein được nghiên cứu kỹ nhất là chaperonin GroEL–GroES, thường gặp ở các loài vi khuẩn. Phức hợp GroEL–GroES ngăn cản sự bám dính những chuỗi polypeptide đồng loại bằng cách nhét riêng rẽ chúng vào trong những cấu trúc dạng túi của nó. Tại đây các chuỗi polypeptide có thể tiếp tục thực hiện quá trình tạo hình của mình mà ko bị dính với các chuỗi khác [11],[12]. Một nghiên cứu từ trước kia của nhóm Ulrich Hartl [13] đã thấy rằng protein Rubisco của vi khuẩn có thể cuốn xoắn chính xác nhanh hơn từ 3 đến 4 lần khi nằm trong túi GroEL-GroES so với việc tạo hình trong một dung dịch kể cả khi ít có khả năng bị bám dính với chuỗi peptide khác. Nhóm này gần đây lại phát hiện [14] rằng cả kích thước và điện tích bề mặt của các túi GroESL cũng đã được tối ưu hóa để tăng tốc quá trình tạo hình của nhiều loại chuỗi polypeptide khác nhau.
3) Khi có mặt ATP hay ADP, các phân tử GroEL và GroES có thể kết hợp với nhau tạp thành phức hệ tạo túi (nanocage). GroES có chức năng như là một chiếc nắp đóng mở để giữ chuỗi peptide nằm yên trong túi khi tạo hình. Bên trong chiếc túi này, những chuỗi peptide tiếp tục cuộn xoắn cho đến khi những amino acid có tính kỵ nước, loại amino acid thường gây bám dính lung tung, đã dược cuộn vào bên trong phân tử protein một cách chặt chẽ. Cái túi này thoạt đầu có tên "túi Anfinsen" với ngụ ý là những chuỗi peptide được cuộn xoắn theo trình tự amino acid của chúng, vì rằng Christian Anfinsen là người thực hiện thí nghiệm chứng tỏ một protein sau khi biến tính có thể tạo hình trở lại. Tuy nhiên, thí nghiệm mới đây [15] cho thấy mô hình này không hoàn thiện, bởi vì tốc độ tạo hình của một vài protein phụ thuộc nhiều vào kích thước tương đối của chuỗi peptide với kích thước túi, và cũng phụ thuộc vào đặc tính bề mặt bên trong những túi GroES-GroEL. Do vậy, phức hệ GroES-GroEL không chỉ là một "dụng cụ" chống bám dính protein mà còn cho phép protein có thể cuộn xoắn nhanh và chính xác hơn.
4) Có khoảng 85 protein khác nhau đã tham gia vào quá trình tạo nên cấu trúc túi bên trong phức hệ GroEL-GroES ở Escherichia coli. Hiển nhiên, đa số (60%) những protein này có kích thước nhỏ (30–50 kilodaltons) và chỉ 14% protein là lớn hơn 50 kDa. Cái túi GroEL–GroES đã được đo là rộng khoảng 85 angstroms (phép đo được thực hiện với 80 túi khác nhau), kích thước này về nguyên tắc là vừa đủ để chứa những protein có kích thước tới 70 kDa. Tuy nhiên, khoảng không gian có thể hơi nhỏ hơn một chút vì có thêm 23 amino acid ở các đầu thừa ra của mỗi tiểu đơn vị GroEL. Việc loại bỏ những đuôi thừa này không làm ảnh hưởng cơ chế hoạt động của túi, và do đó cho phép thay đổi kích cỡ túi. Bằng việc bỏ bớt hoặc thêm vào các đuôi thừa này, Tang et al. [16] đã điều chỉnh thể tích túi cỡ +4% đến -13%, và ghi nhận những ảnh hưởng đến tốc độ tạo hình của 4 loại protein khác nhau có kích thước từ 33 - 50 kDa.
- ^ Ellis, R. J. in Molecular Chaperones and Cell Signalling (eds Henderson, B. & Pockley, G.) 3–21 (Cambridge Univ. Press, 2005)
- ^ Saibil, H. R. & Ranson, N. A. Trends Biochem. Sci. 27, 627–632 (2002)
- ^ Fenton, W. A. & Horwich, A. L. Q. Rev. Biophys. 36, 229–256 (2003)
- ^ Brinker, A. et al. Cell 107, 223–233 (2001)
- ^ Tang, Y. -C. et al. Cell 125, 903–914 (2006)
A. Mogk và B. Bukau (Current Biology 2004 14:R78)[sửa]
1) Việc protein tạo hình sai hoặc kết vón lẫn nhau là một mối đe dọa đến sự tồn tại của tất cả các sinh vật sống. Do đó, tế bào đã phát triển các hệ thống kiểm soát chất lượng protein thông qua 1) những phân tử "chaperone" giúp protein tạo hình lại và 2) các protease để phân hủy các protein sai hỏng cấu hình. Khi nhiệt độ xung quanh tế bào tăng cao, hoạt động của chaperone và protease có thể bị quá tải do lượng protein sai hỏng quá nhiều và hiện tượng kết vón protein lại diễn ra. Hiện tượng này rất quan trọng trong bệnh lý vì có thể dẫn đến những bệnh liên quan hệ thần kinh như Parkinson hoặc prion.
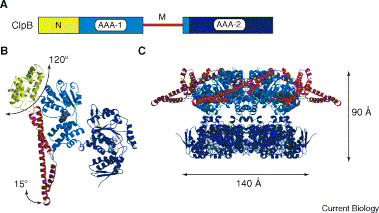
2) Khi protein đã bị kết vón thì coi như đã bị hỏng hoàn toàn vì các phân tử chaperone trước đây chỉ có thể ngăn ngừa việc kết vón và tái tạo những protein sai hỏng cấu hình. Tuy nhiên, trong những năm 1990, Lindquist và cộng sự [1] đã phát hiện một nhóm protein cảm ứng nhiệt Hsp (Heat shock protein) ở nấm men Saccharomyces cerevisiae, gọi là Hsp104. Hsp104 là nhân tố thiết yếu cho quá trình chịu nhiệt, một trạng thái sinh lý cho phép tế bào có thể chống chịu stress sau khi xử lý nhiệt tăng cường. Những nghiên cứu sau này cho thấy cơ chất của Hsp104 là những cục vón protein được tạo ra trong quá trình chịu nhiệt. Những tế bào nấm men mang Hsp104 mất chức năng thì sẽ không còn khả năng làm tan và hoạt hóa lại những protein đã bị kết vón [2]. Chức năng của Hsp104 được tìm thấy và bảo thủ ở các đại diện [[:vi:vi khuẩn|vi khuẩn thực]], thực vật và ty thể. Và những protein đồng dạng với Hsp104 như ClpB, Hsp101 và Hsp78 cũng là những phân tử thiết yếu của phản ứng chịu nhiệt và chống chịu kết vón protein [3-5].
3) Hsp104/ClpB thuộc vào siêu họ protein AAA+, chúng có domain ATPase cũng với các domain tương tác với nhiều loại protein nội bào và protein thuộc Clp/Hsp100. Hai nhóm protein này có độ tương đồng cao về trình tự ở các domain AAA, vùng chức năng quan trọng để thủy phân ATP và tạo phức hệ protein (oligomer hóa). Thông thường, những protein AAA+ thường tạo thành những phức hệ 10 phân tử đồng dạng (homohexamer) có hình chiếc nhẫn. Cấu hình này có thể tháo và lắp dễ dàng và phù hợp với các phức hệ cơ chất phức tạo khác nhau.
References:
- Sanchez, Y. and Lindquist, S.L. (1990). HSP104 required for induced thermotolerance. Science 248, 1112-1115.
- Parsell, D.A., Kowal, A.S., Singer, M.A., and Lindquist, S. (1994). Protein disaggregation mediated by heat-shock protein Hsp104. Nature 372, 475-478
- Mogk, A., Tomoyasu, T., Goloubinoff, P., Rüdiger, S., Roeder, D., Langen, H., and Bukau, B. (1999). Identification of thermolabile E. coli proteins: prevention and reversion of aggregation by DnaK and ClpB. EMBO J. 18, 6934-6949
- Schmitt, M., Neupert, W., and Langer, T. (1996). The molecular chaperone Hsp78 confers compartment-specific thermotolerance to mitochondria. J. Cell Biol. 134, 1375-1386
- Queitsch, C., Hong, S.W., Vierling, E., and Lindquist, S. (2000). Heat shock protein 101 plays a crucial role in thermotolerance in Arabidopsis. Plant Cell 12, 479-492
F.U. Hartl và M. Hayer-Hartl (Science 2002 295:1852)[sửa]
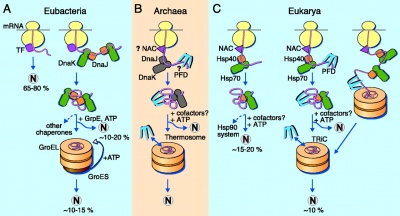
1) To become functionally active, newly synthesized protein chains must fold to unique three-dimensional structures. How this is accomplished remains a fundamental problem in biology. Although it is firmly established from refolding experiments in vitro that the native fold of a protein is encoded in its amino acid sequence, protein folding inside cells is not generally a spontaneous process. Evidence accumulated over the last decade indicates that many newly synthesized proteins require a complex cellular machinery of "molecular chaperones" in addition to the input of metabolic energy to reach their native states efficiently. The various chaperone factors protect nonnative protein chains from misfolding and aggregation, but do not contribute conformational information to the folding process.
2) Spontaneous refolding in vitro is generally efficient for small, single-domain proteins that bury exposed hydrophobic amino acid residues rapidly (within milliseconds) upon initiation of folding. In contrast, larger proteins composed of multiple domains often refold inefficiently, owing to the formation of partially folded intermediates, including misfolded states, that tend to aggregate. Misfolding originates from interactions between regions of the folding polypeptide chain that are separate in the native protein and that may be stable enough to prevent folding from proceeding at a biologically relevant time scale. These nonnative states, though compact in shape, often expose hydrophobic amino acid residues and segments of unstructured polypeptide backbone to the solvent. They readily self-associate into disordered complexes, driven by hydrophobic forces and interchain hydrogen bonding. This aggregation process irreversibly removes proteins from their productive folding pathways, and must be prevented in vivo by molecular chaperones. A certain level of protein aggregation does occur in cells despite the presence of an exclusive chaperone machinery and, in special cases, can lead to the formation of structured, fibrillar aggregates, known as "amyloid", that are associated with diseases such as Alzheimer's or Huntington's disease. Compared to refolding in dilute solution, the tendency of nonnative states to aggregate in the cell is expected to be sharply increased as a result of the high local concentration of nascent chains newly synthesized proteins in polyribosomes and the added effect of macromolecular crowding.
References (abridged):
1. C. Dobson and M. Karplus, Curr. Opin. Struct. Biol. 9, 92 (1999)
2. R. J. Ellis and S. M. Hemmingsen, Trends Biochem. Sci. 14, 339 (1989)
3. M.-J. Gething and J. Sambrook, Nature 355, 33 (1992)
4. F. U. Hartl, Nature 381, 571 (1996)
5. J. Frydman, Annu. Rev. Biochem. 70, 603 (2001)
Những vấn đề tạo hình protein[sửa]
Jeffery W. Kelly (Nature 2005 437:486)[sửa]
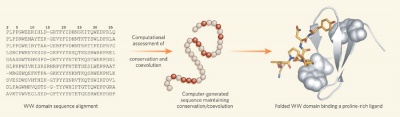
1) The linear sequence of amino acids in a protein specifies its final three-dimensional structure and function[1]. But what molecular information is necessary and sufficient to specify protein form and function? New work[2,3] demonstrates that maintaining the conservation pattern in a protein family, along with a surprisingly small subset of coevolving residues, enables the generation of low-homology sequences that fold and function. The studies indicate that the number of crucial interactions in a protein may be smaller than previously thought -- a boon for those who want to design novel proteins from scratch to fulfill a specific function.
2) The new work studied a large family of protein modules, called the WW domains, that mediate protein-protein interactions by binding to sequences that are rich in the amino acid proline[4]. The researchers aligned 120 WW domains from natural proteins, and looked at the distribution of amino acids that occurs at all of the positions along the polypeptide. By comparing each position against the mean distribution from all proteins, they identified those positions that have been conserved throughout evolution and are therefore likely to have some structural or functional significance. Conservation in these terms means that the amino acid is the same.
3) They next identified amino-acid positions within the conserved set that seem to have evolved in concert. For example, looking at the 74 sequences with glutamate at position 8 allows comparisons with other positions. Position 16 is a "conserved" position, but it exhibits a mean distribution of residues in the 74 Glu8 sequences, revealing that position 16 is not coupled to Glu8. However, some conserved positions are coupled to Glu8 -- that is, they have coevolved. For instance, position 23 has sequence bias in the 74 Glu8 sequences that is distinct from that at the same position in the 120-sequence alignment.
4) Position 8 is statistically coupled (P < 0.05) to just six other sites in the sequence, and a matrix of evolutionary couplings for several positions shows a pattern where a few positions are mutually conserved and most positions interact only weakly, if at all. This result was surprising, and suggested that a computer program might be able to design novel WW domain sequences using conservation and coupling information only, without any information about three-dimensional structure.[5]
References (abridged):
1. Anfinsen, C. B. Science 181, 223-230 (1973) 2. Socolich, M. et al. Nature 437, 512-518 (2005) 3. Russ, W. P., Lowery, D. M., Mishra, P., Yaffe, M. B. & Ranganathan, R. Nature 437, 579-583 (2005) 4. Sudol, M., Chen, H. I., Bougeret, C., Einbond, A. & Bork, P. FEBS Lett. 369, 67-71 (1995) 5. Jager, M., Nguyen, H., Crane, J. C., Kelly, J. W. & Gruebele, M. J. Mol. Biol. 311, 373-393 (2001)
Tạo hình protein và những cụm tương tác kỵ nước[sửa]
The following points are made by Robert L. Baldwin (Science 2002 295:1657):
1) It has been hotly debated whether a hydrophobic collapse precedes or occurs concomitantly with formation of secondary structures at the beginning of protein folding (1). The role of secondary structures in guiding the folding pathway is readily understood because they provide the framework for the final native structure. It has also long been recognized (2) that burial of nonpolar (hydrophobic) side chains out of contact with water provides the major source of the free energy change that drives folding. But compelling models have been lacking for how this burial might produce a "hydrophobic collapse" that initiates folding.
2) The most plausible model has been the "hydrophobic zipper" (3), in which clusters of nonpolar side chains stabilize secondary structures such as a helices or beta hairpins. Examples are known in which such hydrophobic clusters guide the folding process (4,5). The clusters persist until folding is complete and can be visualized in the native structure of proteins. Hydrophobic patches have also been observed in native protein structures at sites where two a helices interact, and there is good evidence that hydrophobic clusters of this kind can guide the folding process.
3) In these examples, hydrophobic clusters and secondary structures are formed concomitantly and produce native-like structures. But hydrophobic clusters have also been found in denatured proteins, under conditions where secondary structures are unstable. Some of these clusters must reflect nonnative interactions because they cannot be found in the structures of the native proteins. It remains unclear how these hydrophobic clusters affect the folding process.
4) Hydrophobic clusters in denatured proteins can be detected by various nuclear magnetic resonance (NMR) probes, including the nuclear Overhauser effect and chemical shifts of side chain protons. Clusters are formed not only in water but also in the presence of the denaturant urea. A particularly useful probe for examining which residues form a hydrophobic cluster in a denatured protein is the transverse relaxation rate, R-2, of a residue's amide group in the peptide backbone, measured by 15N-1H heteronuclear NMR. The value of R-2 expected for a structureless "random coil" can be read from the nearly horizontal plot of R-2 versus residue number. Residues forming a hydrophobic cluster have elevated values that are readily observed in this plot.
References (abridged):
1. S. Akiyama et al., Proc. Natl. Acad. Sci. U.S.A. 99, 1329 (2002) 2. W. Kauzmann, Adv. Protein Chem. 14, 1 (1959) 3. K. A. Dill, K. M. Fiebig, H. S. Chan, Proc. Natl. Acad. Sci. U.S.A. 90, 1942 (1993) 4. K. Zdanowski, M. Dadlez, J. Mol. Biol. 287, 433 (1999) 5. M. E. Hodsdon, C. Frieden, Biochemistry 40, 732 (2001)
Tạo hình protein và bệnh tật[sửa]
The following points are made by Christopher M. Dobson (Science 2004 304:1259):
1) Although there is much to learn about the causes of diseases that involve insoluble protein aggregates, it now appears that they are ultimately linked to a failure of the complex mechanisms that normally ensure that proteins remain in their correctly folded functional states (1-5). Such mechanisms -- including the actions of folding catalysts, molecular chaperones, and degrading enzymes -- normally detect misfolded or damaged proteins and either rescue or destroy them before any harm ensues. If they are not dealt with in this way, these aberrant proteins tend to self-assemble, most notoriously into the highly intractable structures known as "amyloid fibrils" associated with Alzheimer's disease, variant Creutzfeldt-Jakob disease (vCJD), type II diabetes, and many others. Indeed, such fibrils or their precursors appear to cause havoc in any tissues in which they form (4,5).
2) There is increasing evidence that fibrillar aggregates are not esoteric species associated with a small number of proteins, but instead are a generic form of polypeptide structure that results from the dominance of interactions involving the main chain common to all such molecules (5). By contrast, the structures of the normally soluble forms of proteins are dominated by the specific packing of the side chains that distinguish one protein sequence from another. One can therefore think of the amyloid diseases as resulting from the "reversion" of the highly evolved biologically functional forms of peptides and proteins into an alternative and unwelcome structural state that exists as a result of the inherent physicochemical nature of polypeptide chains. The regulatory processes that normally prevent such events -- for example, by maintaining the conditions required for proteins to remain correctly folded -- can fail as a result of many different factors including genetic mutations, ingestion of pathogenic forms of prion proteins, or simply old age.
3) The increasing importance of amyloid diseases to human health has prompted major research efforts to identify the molecular mechanisms that give rise to pathological symptoms and to explore strategies to prevent or reverse them. There have been important developments due to serendipity and large-scale screening programs, but a better understanding of the underlying mechanisms of protein aggregation has accelerated the "rational" design of new drugs. The essence of the mechanism of protein aggregation is that monomeric species bent on aggregation initially form small oligomers that then nucleate the growth of rudimentary fibrillar structures. These species can reorganize and assemble further to produce characteristic long and often twisted thread-like fibrils (4,5). Thus, there are distinct steps in the aggregation process where intervention might be able to prevent or reverse the formation of protein aggregates. In addition, there have been advances in the imaging of amyloid deposits -- even in the human brain -- that use, for example, modified versions of dye molecules that bind specifically to amyloid structures and act as positron emission tomography (PET) tracers. Developments in imaging should enable the biochemical effects of drugs targeted against these diseases to be monitored more effectively in clinical trials.
4) For proteins whose functional state is a tightly packed globular fold, an essential first step in fibril formation is the partial or complete unfolding of the native structure that otherwise protects the aggregation-prone polypeptide backbone. Thus, many of the familial forms of amyloid diseases are associated with genetic mutations that decrease protein stability and promote unfolding. In such cases, one approach to therapy is to find a means of stabilizing the native states of disease-associated variants of amyloidogenic proteins. A series of small-molecule analogs of the hormone thyroxine, the natural ligand of transthyretin (a protein associated with one form of systemic amyloidosis), act in just this way. These small-molecule drugs block the rate at which the disease-associated variants of transthyretin aggregate in vitro. Similarly, specific antibodies raised against lysozyme prevent the formation of amyloid fibrils by pathogenic forms of this antibacterial protein. Moreover, quinacrine, a drug originally developed to combat malaria, limits the replication in cell culture of the pathogenic form of the prion protein associated with CJD. The finding that this molecule interacts with the soluble form of the prion protein suggests that it may stabilize the native state of its aggregation-prone target. Quinacrine has entered clinical trials, and determined efforts are now under way to find more potent forms of this compound.
References (abridged):
1. R. Dalton, E. Check, Nature 427, 5 (2004) 2. A. C. Ghani et al., Proc. R. Soc. London Ser. B 270, 689 (2003) 3. K. R. Merikangas, N. Risch, Science 302, 599 (2003) 4. D. J. Selkoe, Nature 426, 900 (2003) 5. C. M. Dobson, Nature 426, 884 (2003)
References[sửa]
- ^ Fersht, A. R. , Matouschek, A. & Serrano, L. J. Mol. Biol. 224, 771-782 (1992)
- ^ Sadqi, M. , Fushman, D. & Muñoz, V. Nature 442, 317-321 (2006)
- ^ Bryngelson, J. D. , Onuchic, J. N. , Socci, N. D. & Wolynes, P. G. Proteins 21, 167-195 (1995)
- ^ Sabelko, J. , Ervin, J. & Gruebele, M. Proc. Natl Acad. Sci. USA 96, 6031-6036 (1999)
- ^ Muñoz, V. Int. J. Quantum Chem. 90, 1522-1528 (2002)