Review: Một số phương pháp tạo dòng cDNA
Gần 30 năm qua, thư viện cDNA (cDNA library) đã thể hiện hiệu quả rõ rệt trong nghiên cứu sinh học phân tử như: xác định thành phần, cấu trúc của protein được mã hóa bởi đoạn DNA cụ thể, biểu hiện gen của eukaryote trong tế bào prokaryote, nghiên cứu sự biểu hiện của mRNA, và rất nhiều ứng dụng cụ thể khác. cDNA (complementary DNA, copy DNA) được tạo từ mRNA trưởng thành (mature mRNA) sử dụng enzyme Reverse transcriptase. Ở eukaryote, mRNA có đuôi poly Adenine để phân biệt với các loại RNA khác, đồng thời giữ vai trò tạo đọan mồi (primer) trong quá trình phiên mã ngược. Để xây dựng thư viện cDNA nhất thiết phải tạo dòng cDNA tức là tạo số lượng lớn bản sao cDNA dựa vào cấu trúc của mRNA hoặc lượng nhỏ cDNA ban đầu và sự hỗ trợ của thành tựu công nghệ sinh học. Trong nội dung bài viết lần này, sau khi thực hiện công việc tổng hợp cDNA nguồn từ mRNA bằng kỹ thuật RT-PCR, tôi nêu ra ở đây 2 phương pháp tạo dòng cDNA: phương pháp tạo dòng cDNA bằng plasmid với vật chủ là E.coli, và phương pháp tạo dòng cDNA bằng bacteriophage lambda.
Mục lục
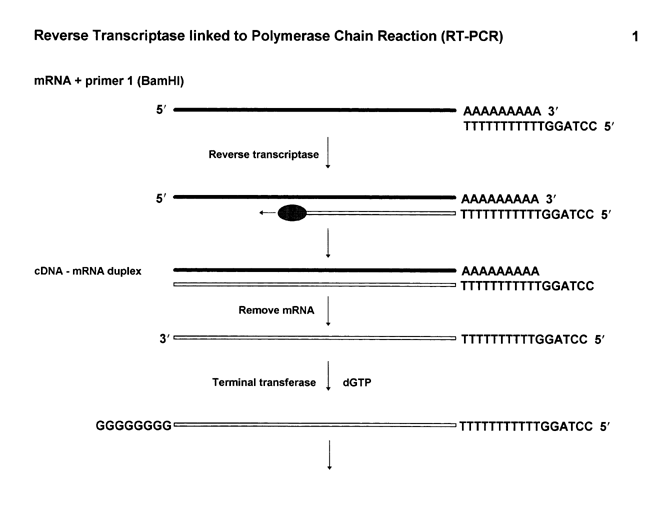
Phương pháp tổng hợp cDNA bằng kỹ thuật RT-PCR (Reverse Transcriptase linked to Polymerase Chain Reaction)[sửa]
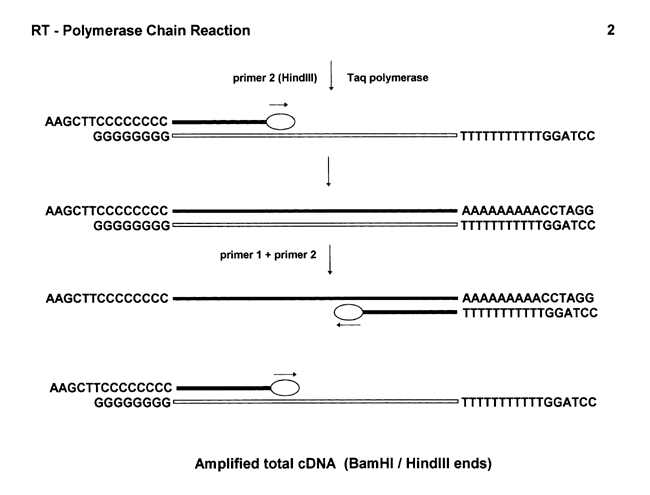
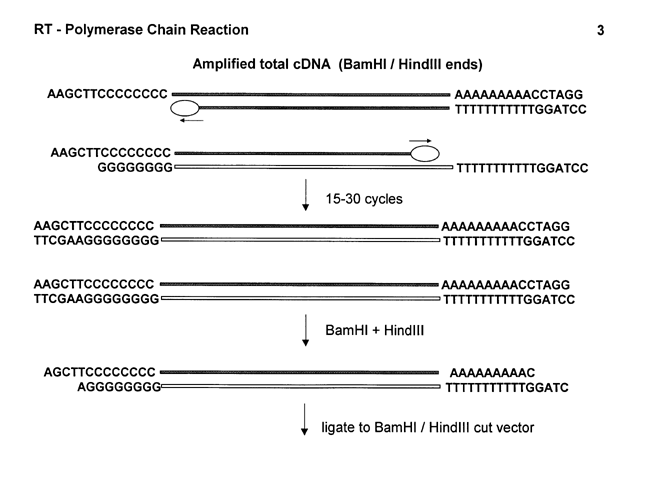
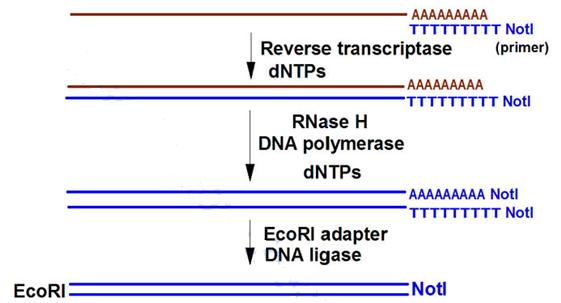
Phương pháp này cơ bản dựa vào phản ứng khuếch đại phân tử (PCR) nhưng có sử dụng enzyme phiên mã ngược, với nguyên liệu ban đầu là phân tử mRNA trưởng thành. Phương pháp này được mô tả qua hình dưới:
Phương pháp này được phát triển để tạo dòng cDNA trong trường hợp lượng mRNA rất ít hoặc lượng mô ít. Hình dưới mô tả một trong nhiều kỹ thuật sử dụng sự kết hợp giữa 2 enzyme reverse transcriptase và Taq polymerase được gọi là RT-PCR. Mục đích của kỹ thuật này là tạo ra đoạn cDNA mang 2 đầu đều có chức năng làm primer và làm vùng cắt hạn chế để chèn vào genome vật chủ phục vụ cho mục đích biểu hiện. Trong ứng dụng này đuôi poly A của mRNA được dùng làm đoạn bổ sung với primer dT được thiết kế mang trình tự của vùng cắt hạn chế cho RE (restriction enzyme) cụ thể. Tương tự như thế, các primer bổ sung tiếp theo dA, dG, hoặc dC mang vùng hạn chế cụ thể nào đó.
Phương pháp tạo dòng cDNA bằng plasmid[sửa]
Sử dụng plasmid để tạo dòng cDNA có kích thước dưới 3 kb với số lượng lớn nhờ vào khả năng sinh sản của vật chủ, thường là E.coli.
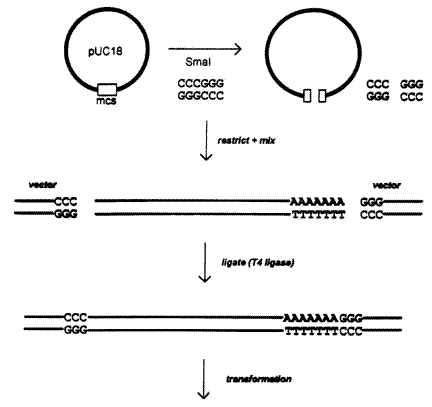
Theo mô tả một cách đơn thuần là khi đưa một đoạn cDNA vào plasmid , người ta chỉ cần cắt plasmid để làm mất cấu trúc vòng đóng, sau đó gắn cDNA vào plasmid bằng enzyme ligase tạo plasmid tái tổ hợp và biến nạp vào E.coli. Nhưng thực tế là, cắt plasmid như thế sẽ tạo ra các đầu bằng (blunt ends), sau khi gắn nối và nhân dòng cDNA sẽ gặp khó khăn để tách lấy cDNA nguyên vẹn dùng cho nghiên cứu tiếp theo.
Vì lí do trên, người ta tạo ra đầu bổ sung giữa plasmid bị cắt và đoạn cDNA đích, hoặc đầu sole ở 1 phía, 2 phía, ở cDNA, plasmid, hoặc cả plasmid và cDNA sẽ có nhiều thuận lợi trong quá trình tạo dòng và tách chiết cDNA.
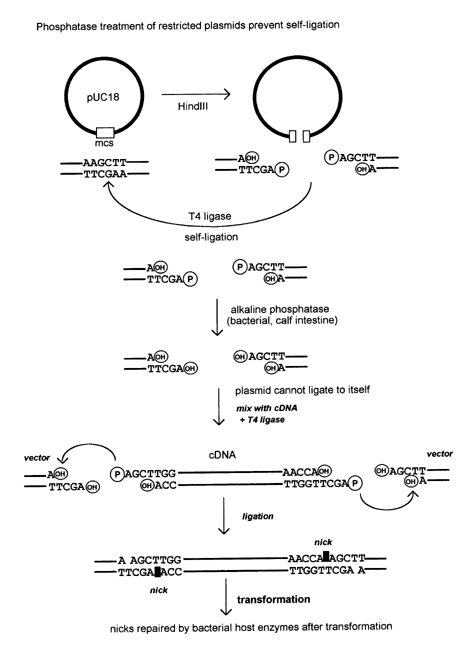
Xử lý plasmid sau khi bị cắt tại vùng hạn chế
Quá trình chèn đoạn cDNA vào plasmid đòi hỏi phải cắt plasmid tại một vùng hạn chế, sau khi cắt thì các plasmid có thể tự gắn nối (self-ligation) lại theo cơ chế bổ sung giữa các nucleotide ở 2 mạch bổ sung. Để tạo hiệu suất cắt hạn chế cao, tránh hiện tượng tự gắn nối của plasmid, có 2 biện pháp giải quyết vấn đề này: (1) Cắt Plasmid bằng 2 enzyme cắt hạn chế khác nhau tại vùng cắt hạn chế (MCS) sẽ tạo các đầu bằng, sole khác nhau; (2) Loại bỏ phospho ở đầu 5’ mỗi sợi của plasmid sau khi cắt hạn chế, được mô tả ở hình dưới:
1.Gắn đuôi đồng trùng hợp
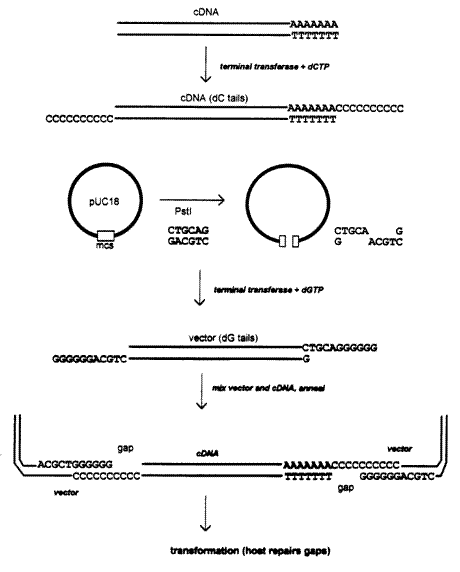
Gắn đuôi dA-dT hoặc dG-dC với số lượng dN khoảng vài chục đến hơn một trăm nucleotide vào 2 đầu của cDNA hoặc của plasmid sẽ hình thành một đoạn đặc trưng là đuôi đồng trùng hợp. Và thực tế, người ta chủ yếu dùng đuôi trùng hợp dG-dC vì nó tạo vùng nhận biết cho một số RE, trong khi nếu dùng đuôi trùng hợp dA-dT thì không có phương thức thích hợp để cắt. Để tạo đuôi đồng trùng hợp này, người ta dùng enzyme teminal transferase, là một DNA polymerase độc lập có tác dụng xúc tác gắn đuôi đồng trùng hợp (homopolymer tail) vào đầu 3’ của chuỗi DNA.
Tạo đuôi đồng trùng hợp, chủ yếu là dC và dG vào các đầu 3’ ở 2 chuỗi DNA của plasmid và của 2 sợi cDNA, sau đó ủ chúng với nhau để gắn đoạn cDNA vào vector plasmid mà không cần dùng enzyme gắn nối nào. Sau khi chèn, ở vector plasmid tái tổ hợp xuất hiện khoảng trống (gap), nhưng sẽ được lấp đầy bởi các enzyme trong tế bào chủ tạo dòng.
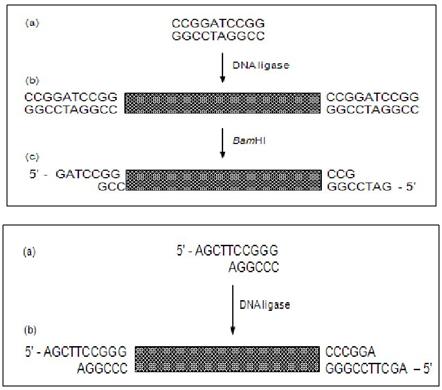
2.Sử dụng adapter hoặc linker tạo đầu dính (cohensive end)
Linker là một sợi đôi nucleotide đầu bằng (blunt end) mang một hay nhiều vùng nhận biết hạn chế, adapter là một oligonucleotide có một đầu bằng (gắn với cDNA sợi đôi) và một đầu dính (gắn với đầu tương ứng ở vector) là vùng nhận biết hạn chế của một RE cụ thể.
Có thể hạn chế sự tái tạo lại vòng của plasmid vector (không tái tổ hợp) bằng cách xử lý các vector được cắt bằng enzyme phosphatase (calf intestinal alkaline phosphatase-CIAP) trước khi thực hiện phản ứng gắn với cDNA. Trong trường hợp các phân tử cDNA mang các adapter đã được phosphoryl hóa (phosphorylation) có thể tạo thành các phân tử mạch vòng đóng bằng liên kết cộng hóa trị (dạng không thể tạo dòng) hoặc các phân tử mạch thẳng dạng khảm (dạng không mong muốn) trong suốt phản ứng gắn tuần tự với sự có mặt của vector DNA đã được dephosphoryl hóa.
Sử dụng adapter hiệu quả hơn linker vì không phải dùng RE để cắt hạn chế. Trong quá trình gắn nối linker, adapter cần thực hiện một dung tích tối thiểu để duy trì nồng độ của chúng cao, thường gấp 100 lần nồng độ đầu tận cùng của cDNA.
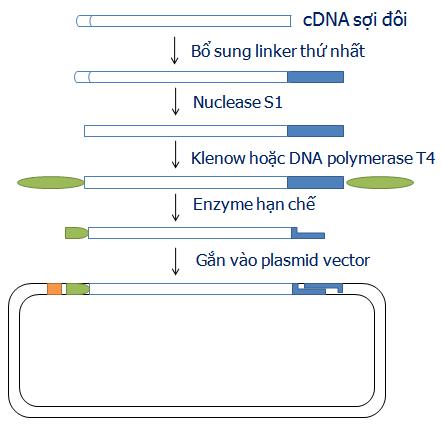
- Để tránh sự gắn nối của cDNA sau khi được gắn adapter hoặc linker, ngoài biện pháp dephosphoryl hóa nêu ở trên, có thể dùng phương pháp bổ sung tuần tự linker vào lần lượt từng đầu của cDNA sợi đôi. Linker đầu tiên được gắn vào một đầu của sợi đôi cDNA chưa bị loại bỏ vòng kẹp tóc (hairspin loop); mục đích của việc giữ vòng kẹp tóc lại là để linker chỉ gắn vào một đầu của cDNA (đầu bằng).
3.Tạo dòng bằng primer-adapter
Primer-adapter là một trình tự nucleotide mang một poly Thymine được gắn thêm chuỗi nucleotide là một vùng nhận biết hạn chế nào đó. Primer-adapter này được dùng ngay từ quá trình tổng hợp cDNA từ mRNA, như vậy primer-adapter này vừa có khả năng bắt cặp với trình tự polyA của mRNA, vừa mang luôn một mạch của vùng nhận biết hạn chế. Sau khi tổng hợp ra 2 mạch mRNA-DNA, rồi cDNA sợi đôi thì chúng sẽ chứa sẵn một vùng cắt hạn chế nên sẽ không cần phải gắn thêm adapter hay linker nữa.
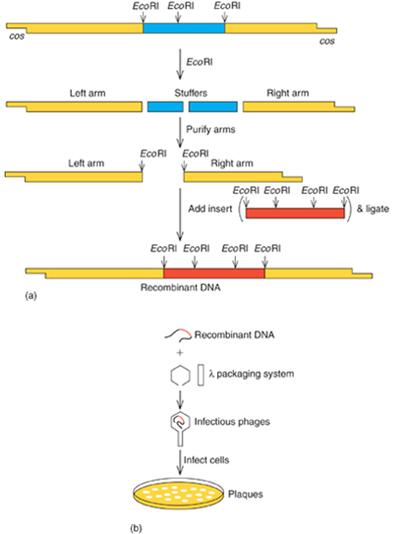
Tạo dòng cDNA với vector bacteriophage lambda[sửa]
Vector lambda mang vùng đệm (suffer) hay còn gọi là vùng trung tâm có chiều dài bằng 1/3 chiều dài của toàn bộ DNA, vùng này không quan trọng nên nếu cắt bỏ sẽ không ảnh hưởng đến khả năng sinh sản của phage lambda. Vì thế người ta loại bỏ vùng này và thay bằng đoạn cDNA nói riêng, đoạn DNA nói chung vào vị trí đó. DNA của phage lambda mang một số vùng cắt hạn chế, các thế hệ lambda cải tiến mang nhiều vùng nhận biết hạn chế hơn. Trong tạo dòng cDNA, thường dùng bacteriophage lambda gt10, gt11, và lambda ZAP I, II là thế hệ bacteriophage lambda mới.
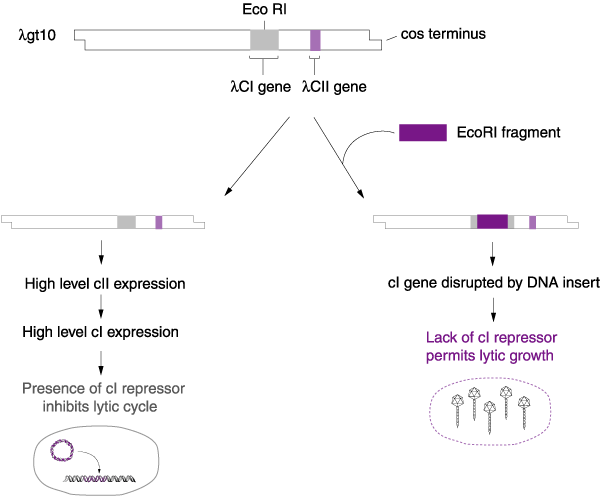
1.Vector lambda gt10
Lambda gt 10 là vector được thiết kế để nhận đoạn DNA ngoại lai trong vùng mã hóa gen ức chế cI2. Các vector này mang gen cI, giống như bacteriophage lambda, nó tạo thành các vết tan mờ đục trên hầu hết các chủng E.coli. DNA của lgt 10 mang vị trí nhận biết đơn EcoRI nằm trong vùng mã hóa gen cI, sản sinh ra các bacteriophage tái tổ hợp và tạo thành các plaque màu sáng, dễ phân biệt với các plaque mờ đục của lgt bố mẹ.
DNA của lgt 10 có kích thước khoảng 43 kb và có thể nhận được các đoạn DNA ngoại lai có kích thước lên tới 7,6 kb. Các thư viện cDNA được xây dựng trong lgt10 luôn chứa hỗn hợp các bacteriophage tái tổ hợp và không tái tổ hợp. Hai loại bacteriophage này có thể phân biệt kiểu hình nhờ vào khả năng tạo các plaque. Hình dưới mô tả việc chèn cDNA vào DNA của lgt 10 và phân biệt các chủng sau khi tái tổ hợp.
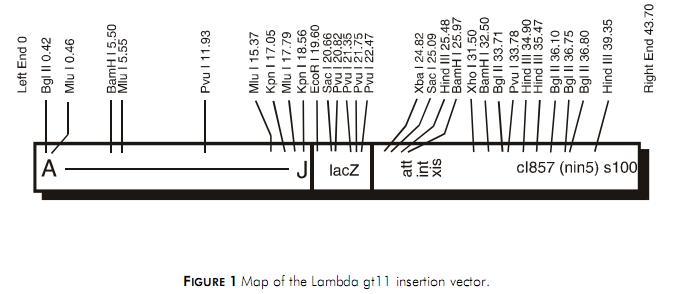
2.Vector lgt 11
Lgt 11 là vector biểu hiện, mang bản sao của gen lacZ của E. coli, có vị trí nhận biết đơn EcoRI nằm ở vùng ngược hướng 53 bp của codon kết thúc dịch mã của gen lacZ. DNA ngoại lai có kích thước xấp xỉ 7,2 kb có thể được gắn ở vị trí này. Các chuỗi mã hóa gắn trong khung đọc theo hướng chính xác sẽ được biểu hiện để sản xuất các protein dung hợp (fusion protein) có đầu tận cùng amino chứa các chuỗi β-galactosidase và đầu tận cùng carboxyl chứa mạch polypeptide ngoại lai. Một số protein dung hợp này sẽ thể hiện các yếu tố quyết định kháng nguyên (epitopes) được phát hiện bởi khả năng phản ứng với các kháng thể của chúng.
Hình dưới là mô hình cấu trúc DNA của lgt 11:
3.Vector lambda ZAP
Vector lZAP mang vùng tạo dòng cùng hướng với promoter lacZ của E. coli. Đoạn cDNA dài 10 kb có thể được gắn vào trong các vị trí này và biểu hiện hoặc trong vi khuẩn được xâm nhiễm hoặc trong các thể tiềm tan được cảm ứng, như mô tả ở lgt 11. Hơn nữa, các protein dung hợp lZAP có thể được biểu hiện từ các plasmid có số lượng bản sao lớn. Vector lZAP có một số đặc điểm như sau:
- Có sáu vị trí RE trong vùng tạo dòng được sử dụng để tạo dòng định hướng cDNA, trong đó có một vị trí cắt EcoRI tương tự như lgt 11.
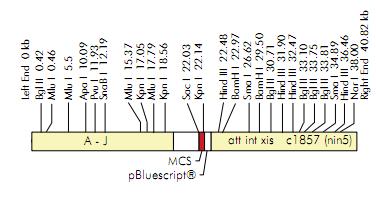
- Có các promoter của bacteriophage T3 và T7 nằm cạnh vùng tạo dòng. Hình dưới là bản đồ các vùng cắt hạn chế của lZAP II.
Tham khảo[sửa]
- N.H.Lộc, 2004. Công nghệ DNA tái tổ hợp. NXB ĐHQG tpHCM
- Minglin Lang, Yuxiu Zhang. PCR-Enriched cDNA Pool Method for Cloning of Gene Homologues. Plant Molecular Biology Reporter, 2005, 23: 219–226
- S. Rajesh, R. Balasaraswathi. Synthesis and Cloning of cDNA Encoding an Antiviral Protein from the Leaves of Bougainvillea spectabilis Willd. (Nyctaginaceae). World Journal of Agricultural Sciences, 2005, 1: 101-104
- Robert F. Weaver. Lecture powerpoint to accompany: Molecular Biology (4th Edition). The McGraw-Hill Companies, 2007
- Vijay M. Chauthaiwale. Bacteriophage lambda as a Cloning Vector. Microbiological Reviews, 1992, Vol. 56, No. 4, p. 577-591